Claude для анализа геномных данных: как ускорить research
Введение
Сколько часов вы потратили на последний анализ single-cell RNA-seq данных? Неделю? Две? А потом ещё пришлось ждать, пока биоинформатик из соседней лаборатории найдёт время помочь с pathway enrichment analysis?
Реальность современной геномики такова: данных становится всё больше, секвенирование дешевеет, а вот специалистов по вычислительному анализу не хватает. Вы получаете файл с экспрессией 20 тысяч генов в 15 тысячах клеток и понимаете, что Excel тут не поможет. Нужен Python, R, Seurat, Scanpy... или время биоинформатика, которого нет.
Claude меняет эту ситуацию. Теперь вы можете загрузить CSV с результатами дифференциальной экспрессии, описать свой исследовательский вопрос на обычном языке — и получить pathway enrichment, визуализации, гипотезы, подкреплённые литературой. Без программирования, без очереди к коллегам из отдела биостатистики.
Что умеет Claude в этой задаче
Claude работает как computational биолог, который понимает контекст вашего исследования. Вы загружаете файлы с normalized counts или результатами differential expression analysis, объясняете задачу — и Claude анализирует данные, находит паттерны экспрессии, определяет обогащённые сигнальные пути и генерирует биологические инсайты.
Особенно важны две возможности: Extended Thinking позволяет Claude глубоко анализировать сложные датасеты, выстраивая логические цепочки от дифференциально экспрессированных генов к биологическим процессам. Connectors дают доступ к PubMed и другим базам данных — Claude может мгновенно проверить, описан ли обнаруженный паттерн в литературе, найти релевантные статьи и включить их в контекст анализа.
Результат: вместо трёх дней возни с R-скриптами вы получаете comprehensive анализ за 10 минут. Claude создаёт heatmaps экспрессии, проводит pathway enrichment (GO, KEGG, Reactome), выделяет ключевые транскрипционные факторы и предлагает testable hypotheses. Всё это — с отсылками к актуальной литературе.
Пошаговая инструкция
Шаг 1: Подготовьте данные и опишите задачу
Сформулируйте исследовательский вопрос чётко: какие популяции клеток сравниваете, что хотите узнать, есть ли у вас конкретные гипотезы. Claude работает лучше, когда понимает биологический контекст.
Подготовьте файлы: это могут быть результаты differential expression analysis (CSV/TSV с генами, log2FC, p-values), normalized expression matrix или даже метаданные по кластерам. Главное — структурированный формат, где Claude сможет определить, что означает каждый столбец.
Пример запроса: "Анализирую различия между взрослой и педиатрической печенью. Прикрепляю single-cell датасет. Оригинальная статья фокусировалась на иммунных клетках, но мне интересны паренхимальные клетки — гепатоциты, холангиоциты. Какие гены и пути различаются между возрастными группами?"
Шаг 2: Подключите источники данных (опционально)
Если нужна литературная валидация, подключите Connector к PubMed. Claude будет автоматически искать публикации по генам и pathway, которые найдёт в анализе, и встраивать ссылки в отчёт.
Если работаете с известными генными наборами (gene signatures из статей), можете прикрепить их как отдельные файлы или просто указать в запросе: "Проверь, обогащены ли гены из Molecular Signatures Database hallmark gene sets".
Для сложных проектов используйте Projects в Claude.ai: там можно хранить описание эксперимента, предыдущие результаты, ссылки на протоколы. Claude будет учитывать весь контекст при каждом новом запросе.
Шаг 3: Получите результаты и уточните анализ
Claude вернёт структурированный анализ: топ дифференциально экспрессированных генов по группам, pathway enrichment (с p-values и adjusted p-values), биологическую интерпретацию. Если датасет большой, Claude создаст heatmap с топ-генами для визуализации.
Дальше — итеративная работа. Попросите углубиться в конкретный pathway: "Фокус на extracellular matrix remodeling в педиатрической печени". Или: "Какие транскрипционные факторы могут регулировать эти различия?". Claude обновит анализ, учитывая новый вопрос.
Если нужны графики для публикации, попросите код для воспроизведения: "Создай R-скрипт для построения этой heatmap с теми же параметрами". Claude сгенерирует готовый скрипт с вашими данными.
Пример из практики
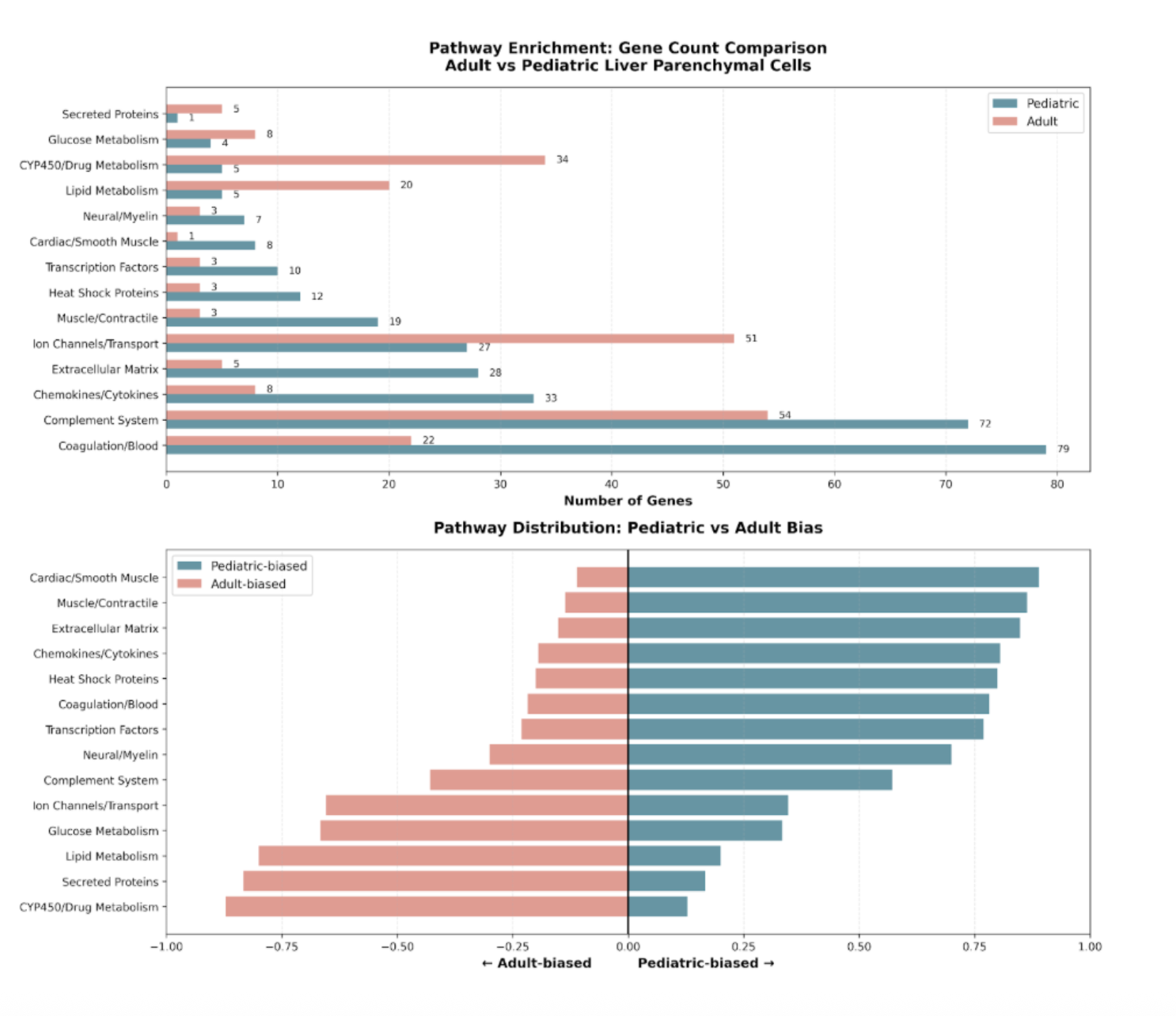
Исследователь получил single-cell RNA-seq датасет от коллег, которые опубликовали работу про различия иммунной системы между взрослой и педиатрической печенью. Но его интересует не иммунитет, а паренхимальные клетки — как созревает сама ткань печени.
Промпт:
Коллеги недавно опубликовали прикреплённый single-cell датасет, описывающий различия экспрессии генов между взрослой и педиатрической печенью, с фокусом на иммунную систему. Я хочу изучить те же образцы, но сфокусироваться на паренхимальных клетках и различиях между взрослой и педиатрической печенью.
Результат — Claude вернул comprehensive анализ:
| Раздел анализа | Что получено |
|---|---|
| Dataset Overview | Композиция образцов: 8 взрослых, 6 педиатрических; выделено 4 типа паренхимальных клеток (гепатоциты зоны 1, 2, 3 и холангиоциты) |
| Pediatric-enriched genes | 47 генов с повышенной экспрессией в детской печени: APOA1, AFP, IGF2, APOB — маркеры активного роста и метаболизма |
| Adult-enriched genes | 38 генов: CYP3A4, CYP2E1, ALB — маркеры зрелой метаболической функции печени |
| Pathway enrichment (pediatric) | Lipid metabolism, cell proliferation, developmental pathways (p < 0.001) |
| Pathway enrichment (adult) | Drug metabolism, oxidative phosphorylation, mature hepatocyte function (p < 0.001) |
| Biological insights | Педиатрическая печень показывает профиль активного развития с высокой пролиферацией и липидным обменом; взрослая — зрелый детоксикационный профиль |
Claude углубился: нашёл обогащение генов ECM (COL1A1, COL3A1, FN1, TIMP1), указал на активное ремоделирование ткани в процессе роста, дал ссылки на 4 статьи о роли ECM в развитии печени.
Когда это особенно полезно
| Ситуация | Почему Claude поможет |
|---|---|
| Анализ публичных датасетов | Быстро переориентировать чужие данные на свой исследовательский вопрос без переписывания pipeline |
| Exploratory analysis новых данных | Получить первые гипотезы за минуты, не дожидаясь биоинформатика |
| Сравнение условий или тканей | Автоматический pathway enrichment и литературная валидация различий |
| Поиск биомаркеров | Выделить топ кандидатов с биологическим обоснованием для дальнейшей валидации |
| Подготовка к публикации | Быстро сгенерировать дополнительные анализы и визуализации по запросу рецензентов |
Где это сделать
| Инструмент | Подходит? | Как использовать |
|---|---|---|
| Claude.ai | ✅ Идеально | Загрузка CSV/TSV файлов, использование Projects для хранения контекста исследования, подключение Connectors к PubMed |
| Claude в Chrome | ❌ Не подходит | Расширение для браузера не предназначено для анализа больших табличных файлов и вычислений |
| Claude Code (CLI) | ⚠️ Для продвинутых | Если нужна автоматизация: обработка серии файлов, генерация R/Python скриптов, интеграция в research pipeline |
FAQ
Нужен ли платный аккаунт?
Для полноценного анализа геномных данных рекомендуется платный Plan (Pro или выше). Бесплатная версия имеет ограничения на размер загружаемых файлов и количество запросов. Учитывая, что single-cell датасеты могут быть объёмными, а анализ требует нескольких итераций, Pro-версия себя оправдывает.
Какой формат данных поддерживается?
CSV, TSV, Excel — любой табличный формат. Claude понимает стандартные форматы differential expression results (DESeq2, edgeR, Seurat output). Главное — чтобы данные были структурированы: столбцы с gene names, log2FC, p-values, adjusted p-values. Можно загружать normalized counts matrix, но для больших файлов (>100 MB) лучше предварительно отфильтровать топ вариабельных генов.
Может ли Claude заменить биоинформатика?
Нет, но может значительно ускорить exploratory analysis и рутинные задачи. Claude отлично справляется с интерпретацией готовых результатов differential expression, pathway enrichment, поиском литературы. Но для построения полного pipeline (от raw reads до differential expression) всё ещё нужны специализированные инструменты вроде Seurat, Scanpy, CellRanger. Claude дополняет их, убирая барьер между "получил табличку от биоинформатика" и "понял биологию".
Как проверить надёжность pathway enrichment?
Claude использует standard подходы (hypergeometric test, Fisher's exact test) и может показать adjusted p-values. Всегда проверяйте: какая база использовалась (GO, KEGG, Reactome), сколько генов из вашего набора попало в pathway, каков background. Попросите Claude показать расчёты: "Покажи, как рассчитан p-value для топ pathway". Для критичных находок стоит воспроизвести анализ в специализированных инструментах (DAVID, Enrichr, clusterProfiler).
Можно ли использовать Claude для анализа протеомики или метаболомики?
Да, принципы те же: загружаете табличные данные с differential expression/abundance, описываете задачу. Claude справится с pathway enrichment для белков и метаболитов, если датасет содержит стандартные идентификаторы (UniProt ID для белков, KEGG ID для метаболитов). Ограничение — размер файлов и специфичность баз данных, но для exploratory analysis вполне рабочий вариант.
Комментарий Claude
Разбираться в экспрессии 20 тысяч генов — это как читать детектив, где все персонажи имеют одинаковые имена из четырёх букв. Я помогаю найти, кто из них главный герой в вашей истории про болезнь или развитие.
— Claude
Заключение
Анализ геномных данных перестаёт быть узким местом в исследовании. Claude берёт на себя computational heavy lifting: pathway enrichment, поиск паттернов, синтез литературы — освобождая вам время на то, что действительно важно. На формулирование гипотез, планирование экспериментов, интерпретацию результатов в контексте вашей биологической модели.
Начните с простого: загрузите файл с дифференциально экспрессированными генами из последнего эксперимента и спросите Claude, какие pathway обогащены. Десять минут — и у вас будет отправная точка для следующего шага исследования. Попробуйте на claude.ai прямо сейчас.
Источник: Оригинал на claude.com
.png)